2. 江苏赞奇科技股份有限公司, 常州, 213022
2. Cudatec Development Co., Ltd, Changzhou, 213022, China
分子对接是用来预测分子之间相互作用和相互识别的模式,而蛋白质分子对接是预测蛋白质受体和蛋白质配体的结合匹配模式[1]。通常,生物学实验是探究分子之间的相互作用的直接手段。但通过实验直接研究蛋白质相互作用并不容易,获得蛋白质复合物晶体的生物实验条件比较严苛,并且实验的成本也非常昂贵,这使理论模拟方法得到了越来越多的重视和应用。分子对接方法在研究分子间相互作用领域有着广泛的应用,可减少单纯实验带来的种种困难,为实验者们提供重要的理论依据。
分子对接主要分为3种,分别是刚性对接(Rigid docking),半柔性对接(Semi flexible docking)和柔性对接(Flexible docking)[2-3]。柔性对接指的是在对接过程中,受体和配体都发生构象的改变,这种对接在于精度上得到了很好的保证,但是随之产生的负面效应是计算量的大幅增加,而刚性对接则恰恰相反。本文主要采用的是半柔性对接,即在保证受体构象不发生变化的情况下,配体的构象发生一定的改变,在计算效率和精度上都能达到相应的要求。
现在被广泛使用的分子对接软件主要有:AutoDock, Molegro Vitual Docker, 3D_Dock, DOCK以及FlexX等[4]。其中,3D_Dock程序具有很大的优越性[5],能够快速进行全局对接和复合物结构数据的筛选。但是,随着对接数据量的不断增大以及计算机硬件设备及处理能力的不断更新,传统的3D_Dock程序已经不能胜任目前的情况。特别是针对半柔性对接计算量比较大,原有的计算方法和并行策略都明显需要改进。
NVIDIA公司发布的统一计算设备架构(Compute unified device architecture,CUDA)[6-7],让GPU作为计算单元得到了强有力的开发。GPU作为图形处理器具有超强的浮点运算能力,而将GPU从传统的图形处理器变为计算单元来使用,最大化地利用其浮点运算能力,有可能解决上述分子对接面临的问题。如何将GPU和3D_Dock程序结合起来是本文的研究重点。
本文在原有3D_Dock程序基础上进行改进。首先,在CPU上,基于FFTW3对原有的程序进行算法升级和程序改进。然后,在GPU上,基于CUFFTW和CUFFT对改进后的程序进行再升级。通过3种方法对程序进行改进和算法升级,用改进程序对PDB code为1PPE的测试蛋白进行结合态和自由态对接,结果验证了程序的正确性并给出了改进后程序的加速比。
1 3D_Dock主要流程分子对接首先要对受体和配体分子结合模式进行全空间搜索[8-9],但是,蛋白质的自身特点决定了空间搜索的困难,即蛋白质对接所产生的复合物数量十分庞大。传统的用于分子对接的空间搜索算法主要有遗传算法、模拟退火算法、快速傅里叶变换算法(Fast Fourier transform,FFT)、片段生长法以及蒙特卡罗算法等。3D_Dock程序主要采用FFT算法来进行复合物构象的计算,只要所需评价函数满足∑ijpiqj的形式,就可以通过FFT算法进行程序的加速,提高采样速度。其主要理论依据在于通过离散函数表现分子特性,具体的流程如下:
假设需要进行对接的受体和配体分子的特性,以及通过对接得到的复合物的特性都通过格点表现出来,则可以通过格点特性来求解特性匹配值。匹配值计算公式如下
| $ {\overrightarrow c _{\alpha , \beta , \gamma }} = \sum\limits_{l = 1}^N {\sum\limits_{m = 1}^N {\sum\limits_{n = 1}^N {{{\overrightarrow a }_{l, m, n}} \cdot {{\overrightarrow b }_{l + a, m + \beta , n + \gamma }}} } } $ | (1) |
式中:
步骤1 确定受体分子和配体分子的投影离散函数
步骤2 分别对投影离散函数,
步骤3 求频域的点乘。
步骤4 根据步骤3得到了所需的复合物结构数据Co, p, q,但是得到的值为频域下的值,因此,下一步需要进行的是傅里叶逆变换。
主要计算公式如下
| $ A_{o, p, q}^ * = {\{ {\rm{FFT}}({\overrightarrow a _{l, m, n}})\} ^*} $ | (2) |
| $ {B_{o, p, q}} = {\rm{FFT}}({\overrightarrow b _{l, m, n}}) $ | (3) |
| $ {C_{o, p, q}} = A_{o, p, q}^ * \cdot{B_{o, p, q}} $ | (4) |
| $ {\overrightarrow c _{\alpha , \beta , \gamma }} = {\rm{IFFT}}({C_{o, p, q}}) $ | (5) |
式(2)为对
经过式(5)计算后得到复合物的相关特性匹配值,根据排序筛选,取得该轮计算的极小值并进行保存,至此便完成了一轮计算。接下来通过改变配体的不同角度继续进行对接,重复以上步骤,得到一系列对接复合物结构。需要特别注意的是,因为是半柔性对接,即受体在构象上不发生变化,因此受体只需要进行一次傅里叶变换即可。完成所有对接情况后,对保留下来的数据继续进行排序,得到打分项最高的复合物结构。
2 优化方案介绍 2.1 基于FFTW的优化方案一3D_Dock的核心程序是FFTW。FFTW主要有两个经典版本FFTW2和FFTW3,但是FFTW3并不兼容FFTW2,而原程序的版本为FFTW2[10-11]。因此,需要有在CPU上优化程序的方案,即将原来FFTW2的函数全部升级为FFTW3的函数。优化方案一基于CPU,将原程序的FFTW2进行升级为FFTW3,其主要流程如下:
步骤1 准备工作:主要涉及内存分配、默认参数的设置、角度计算、原子带电量设定、离散后网格划分、动静态分子结构的读入,以及建立傅里叶变换和逆变换策略。
步骤2 将静态分子进行平面化和离散化,动态分子先采用分子动力学模拟生成3种构象,然后进行离散化。
步骤3 分别将静态分子和动态分子得到的网格数据进行傅里叶变换,并进行点乘计算,得到复合物投影离散函数,并对其进行傅里叶逆变换,得到复合物相关函数。
步骤4 对得到的复合物相关函数进行匹配因子计算,并进行打分排序,筛选出最好的3个结果进行保留。
步骤5 将动态分子进行旋转,重复步骤3,4,直到所有旋转结束。
步骤6 将得到的所有结果进行排序,并写入文件中,上限为10 000个数据。
步骤7 按上述步骤完成受体与另外两个动态分子构象的对接测试。
2.2 基于CUFFTW的优化方案二随着GPU技术的不断革新,NVIDIA公司提出了CUDA架构,同时也针对傅里叶变换发布了两种加速方案,一种是CUFFT加速,另一种是CUFFTW加速。
CUFFTW和CUFFT的区别主要在于,CUFFTW是针对FFTW库开发的,可以最快速、最便捷地为使用FFTW库(方案一采用的加速方法)的用户带来GPU的加速效果,即只要原程序采用的是FFTW库,那么通过CUFFTW可以直接在GPU端进行运行,不需要太多的改动。而CUFFT则不同,它是针对所有傅里叶变换开发的,是一种完全的CUDA接口。
优化方案二在优化方案一的基础上,采用CUFFTW通过部分CUDA GPU接口进行加速。换言之,方案二是将原来在CPU端运行的程序移植到GPU端运行,通过CUDA GPU架构来对方案一进行再加速。具体流程与方案一相同,只是运行环境发生了改变。
2.3 基于CUFFT的优化方案三优化方案一虽然程序确实得到了加速,但是加速效果并不理想;优化方案二专门针对FFTW库开发,不是采用纯CUDA GPU接口,若换成其他函数库则无法使用。因此,考虑到CUFFT版本的加速效果,本文又提出优化方案三,即在GPU端进行纯CUDA接口傅里叶变换,将耗时的快速傅里叶变换运算和复合物点乘计算移植到GPU端,将所得结果返回CPU进行筛选排序处理,并利用GPU的高速浮点运算能力加速程序的运行[12-13]。具体运输步骤如下:
步骤1 根据所做的工作制定一个计划。因为本文傅里叶变换是在三维空间进行的,因此需要调用的函数是cufftPlan3d。其他低维情况可以参考CUFFT的官方文档做相应变动。
步骤2 调用相关函数开始执行计划。因为本文所做的是双精度的傅里叶变换和傅里叶逆变换,所以需要调用的函数分别是cufftExecR2C()和cufftExecuC2R()。
步骤3 调用函数cufftDestroy()释放资源并销毁计划。
以一种动态分子构象计算为例,基于CUDA GPU的3D_Dock程序的具体流程如图 1所示。

|
图 1 基于GPU的分子对接流程图 Fig. 1 Flow chart of molecular docking based on GPU |
3 分子对接实例及结果分析 3.1 对接实例
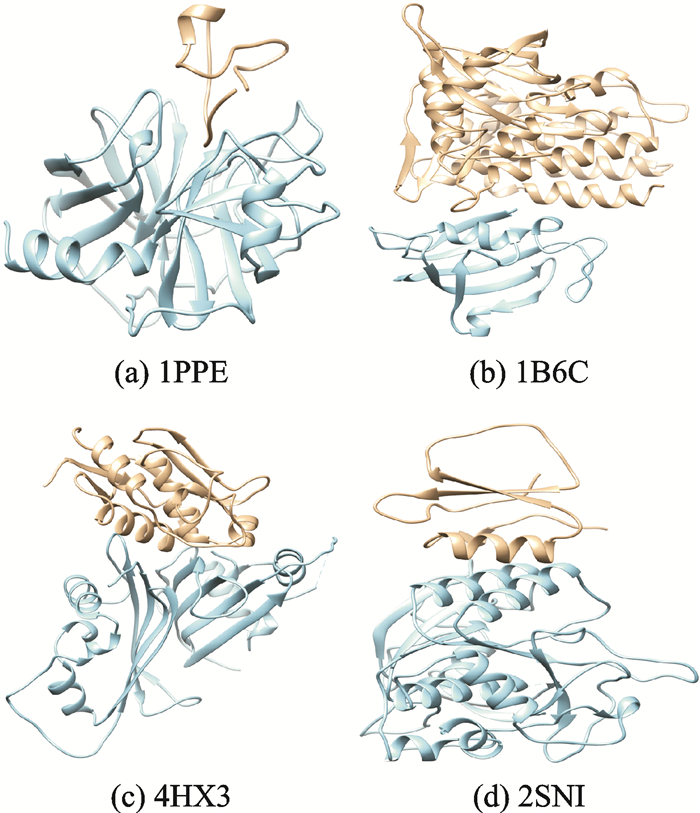
为了测试3D_Dock两种优化方案的有效性以及计算精度,在采用3D_Dock默认对接参数的基础上,将程序应用于PDB code为1PEE,1B6C,4HX3和2SNI的结合态分子对接和自由态分子对接(图 2),求取结合态和自由态模式下的前10名复合物的最小均方根偏差LRMSD值[14-15],同时展示4个体系复合物预测的结构图,比较对接结果与原晶体结构的差异,验证结果的正确性,如表 1所示。
|
图 2 PDB code分别为1PPE,1B6C,4HX3和2SNI的蛋白质分子结构(淡黄色为配体,蓝色为受体) Fig. 2 Molecular structures of 1PPE, 1B6C, 4HX3 and 2SNI(PDB code)(Pale yellow shows the ligand and blue the receptor) |
| 表 1 对接4个体系中具有最小LRMSD值的前10名结合态和自由态结果 Tab. 1 The first ten bound and unbound results of docking instance with the minimum LRMSD value |
评价方式采用蛋白质复合物结构预测竞赛(Critical assessment of predicted interactions, CAPRI)[16-17]组委会的指标。在CAPRI实验中,正确的预测结果分为高精度结构、中等精度结构和可接受结构[18-19]。根据LRMSD划分情况为:当LRMSD小于1 Å时为高精度结构;当LRMSD小于5 Å时为中等精度结构;当LRMSD小于10 Å时为可接受结构。LRMSD表示预测的配体构象与晶体结构中配体结构的均方根偏差。本文选取的标准为LRMSD小于5 Å,即为中等精度下为近天然结构。具体数据来源于CAPRI官方网站: http://www.ebi.ac.uk/msd-srv/capri/。
根据表 1可以看出,结合态结构的LRMSD值要比自由态结构的LRMSD值小。其主要原因是自由态对接主链结构有明显变化,因此自由态的LRMSD值通常要比结合态对接的值大。而结合态结构是直接由复合物晶体结构拆分而来,因此在对接中更容易找到接近晶体结构的复合物构象。图 3是4个PDB结果在结合态结构最低值时(根据表 1结果)对应的对接结果与原晶体结构的对比图,从图中可以确认对接结果的正确性。

|
图 3 4个PDB结合态结果在LRMSD最低值时与原晶体结构对比图 Fig. 3 Comparison of four PDB bound results with the minimum values of LRMSD and the original crystal structure |
3.2 性能分析
为了测试两种方案对于程序的优化效果,对上述4种蛋白均进行了测试,并且加速比基本一致。为便于讨论,选取受体Bovine trypsin和配体CMTI-1 squash inhibitor(PDB code:1PPE)的结合态和自由态对接作为优化测试体系。在保持网格数一定的前提下,通过改变旋转角度步长来比较优化的效果,分别取旋转角度为12°,15°,18°进行测试。表 2,3为3种方案结合态和自由态结构的测试结果,每个测试结果都是配体3种构象的平均值,表中原方案在CPU上运行,采用FFTW2函数库。
| 表 2 基于结合态的4种方案运行时间 Tab. 2 Running time of four optimization schemes based on bound structure |
| 表 3 基于自由态的4种方案运行时间 Tab. 3 Running time of four optimization schemes based on unbound structure |
表 2中优化方案一较原方案在运行时间上有较大幅度的降低。这是由于原方案采用的快速傅里叶变换函数库为FFTW2,而优化方案一采用的函数库为FFTW3。版本2的傅里叶变换主要有fftw.h和rfftw.h两个头文件,分别代表傅里叶变换和傅里叶逆变换;而版本3的傅里叶变换头文件只有1个头文件fftw3.h,即无论是正变换还是逆变换都集中于该头文件。此外,在版本2的傅里叶变换中,原来的实数被双精度浮点型替换,使得精度得到了较大的提高。而且原复数的实部和虚部是在结构体中定义,而在版本3中直接进行宏定义,这样使用起来更加方便和快捷。同时,FFTW2和FFTW3的差别还在于策略的生成与执行。FFTW3将傅里叶变换和反变换的重点从策略的制定转移到策略的执行。如果把计算都放在了策略的生成上面,那么每次生成策略都需要消耗一定的时间,而分子对接工作需要多次傅里叶变换计算,因此采用FFTW2函数库运行时间会大幅增加,而采用FFTW3则可以降低运行时间。优化方案二、三分别是在方案一的基础上进行的再升级,利用CUFFT的两个子库使得部分程序能够在GPU端进行运算,从而降低了程序的运行时间,较之方案一,两者的提速比分别达到3倍和10倍。
由表 3可知,自由态结构和结合态结构一样,在改进后的方案中速度都得到了提高,并且提高幅度相差不大。但是自由态结构的运行时间普遍比结合态结构的运行时间少,这主要是因为部分结合态结构计算所需的格点数要比自由态结构多[20],因此程序运行消耗的时间也相应会有所提高,但是增加的时间并不多,这也一定程度上证实了程序的正确性,以及优化方法的正确性。
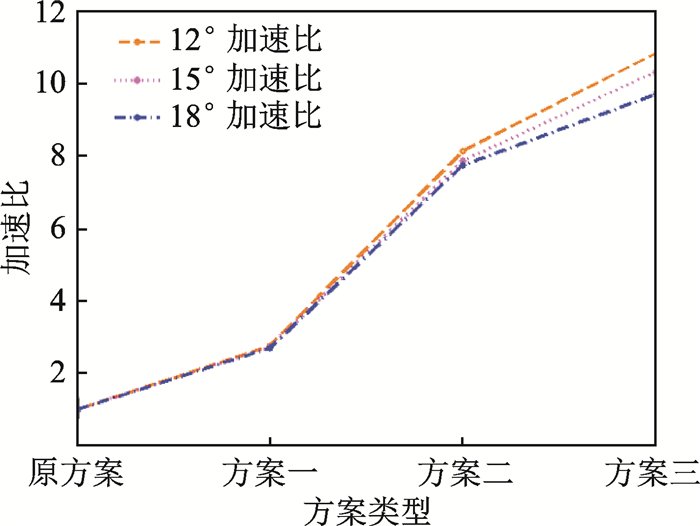
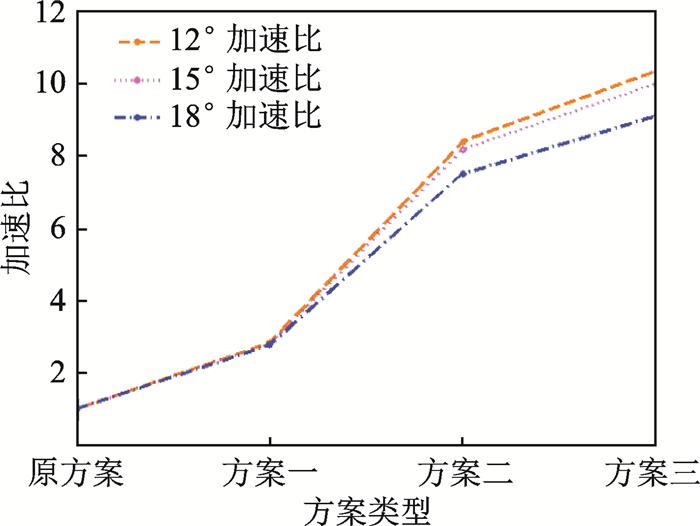
图 4,5是结合态结构和自由态结构中4种方案在旋转角度为12°,15°,18°时的加速比结果,图中所求得的加速比都是配体3种构象测试结果的平均值。
|
图 4 使用4种不同方案时结合态结构加速比对比图 Fig. 4 Accelerated line chart of bound structures using four schemes |
|
图 5 使用4种不同方案时自由态结构加速比对比图 Fig. 5 Accelerated line chart of unbound structures using four schemes |
通过表 2,3及图 4, 5可以得到两方面结论。首先,当旋转步长不断增大时,程序的运行速率得到了提高,这主要是因为当旋转步长增加时,需要旋转采样的次数随之减少,这样所需要的计算量便大幅降低,因此程序运行时间自然减少。其次,如果旋转采样的次数减少,所需要使用的傅里叶变换次数便随之减少,而本文所进行的优化大部分是针对傅里叶变换进行的,一旦傅里叶变换计算次数减少,那么总体的加速比自然会下降。相反,如果旋转步长降低,程序的优化效果则会得到较大的提高,原因在于旋转次数增加,对接采样的角度更加细化,使用快速傅里叶变换的计算次数也会增加。以上结论可以证明本文的优化方案有效,通过对最主要的傅里叶变换算法进行提速,使得程序运行效率得到较大的提高。
4 结束语本文主要针对3D_Dock中傅里叶变换提出了3种不同程度的优化方案,分别是基于CPU的优化和两种基于GPU的优化。从实验结果可以证明较之原程序而言,3种优化方案都提高了程序的运行速度,特别是基于GPU的方案,并行计算的优化更大程度上提高了程序的运行速度。这3种优化方案对于蛋白质结构预测领域[21]有着重要的应用,可以降低计算成本,提高预测效率。
| [1] |
Ritchie D W. Recent progress and future directions in protein-protein docking[J]. Current Protein & Peptide Science, 2008, 9(1): 1-15. |
| [2] |
常珊, 孔韧, 李春华, 等. 基于MPI的分子对接并行算法[J]. 计算物理, 2008, 25(2): 241-246. Chang Shan, Kong Ren, Li Chunhua, et al. Parallel algorithm for molecular docking based on MPI[J]. Chinese Journal of Computational Physics, 2008, 25(2): 241-246. DOI:10.3969/j.issn.1001-246X.2008.02.020 |
| [3] |
王存新, 常珊, 龚新奇, 等. 蛋白质-蛋白质分子对接中打分函数研究进展[J]. 物理化学学报, 2012, 28(4): 751-758. Wang Cunxin, Chang Shan, Gong Xinqi, et al. Progress in the scoring functions of protein-protein docking[J]. Acta Physico-Chimica Sinica, 2012, 28(4): 751-758. DOI:10.3866/PKU.WHXB201202022 |
| [4] |
段爱霞, 陈晶, 刘宏德, 等. 分子对接方法的应用与发展[J]. 分析科学学报, 2009, 25(4): 473-477. Duan Aixia, Chen Jing, Liu Hongde, et al. Application and development of molecular docking method[J]. Journal of Analytical Science, 2009, 25(4): 473-477. |
| [5] |
Moont G, Gabb H A, Sternberg M J E. Use of pair potentials across protein interfaces in screening predicted docked complexes[J]. Proteins, 1999, 35(3): 364-373. DOI:10.1002/(ISSN)1097-0134 |
| [6] |
Sanders J, Kandrot E. CUDA by example:An introduction to general-purpose GPU programming[M]. Upper Saddle River, New Jersey, USA: Addison-Wesley, 2011.
|
| [7] |
Storti D, Yurtoglu M. CUDA for engineers:An introduction to high-performance parallel computing[M]. Upper Saddle River, New Jersey, USA: Addison-Wesley, 2017.
|
| [8] |
Shatsky H J. Flexible protein alignment and hinge detection[J]. Proteins, 2002, 48(2): 242-256. DOI:10.1002/(ISSN)1097-0134 |
| [9] |
Bonvin A M. Flexible protein-protein docking[J]. Current Opinion in Structural Biology, 2006, 16(2): 194-200. DOI:10.1016/j.sbi.2006.02.002 |
| [10] |
赵剑, 阮越, 王嘉松. 数学结构的蛋白质二维数字表达及其应用[J]. 数据采集与处理, 2013, 28(6): 770-776. Zhao Jian, Ruan Yue, Wang Jiasong. 2-D numerical representation of protein based on mathematical structure and its application[J]. Journal of Data Acquisition and Processing, 2013, 28(6): 770-776. DOI:10.3969/j.issn.1004-9037.2013.06.011 |
| [11] |
龚新奇, 刘斌, 常珊, 等. 蛋白质复合物结构预测的集成分子对接方法[J]. 中国科学(C辑):生命科学, 2009, 39(10): 963-973. Gong Xinqi, Liu Bin, Chang Shan, et al. Integrated molecular docking method for prediction of protein complex structure[J]. Chinese Science (Series C):Life Sciences, 2009, 39(10): 963-973. |
| [12] |
吴奎, 宋彦, 戴礼荣. 基于CUDA的GMM模型快速训练方法[J]. 数据采集与处理, 2012, 27(1): 85-90. Wu Kui, Song Yan, Dai Lirong. CUDA based fast GMM model training method and its application[J]. Journal of Data Acquisition and Processing, 2012, 27(1): 85-90. DOI:10.3969/j.issn.1004-9037.2012.01.014 |
| [13] |
李正夫, 王希诚, 郭权. CUDA下受体评分网格生成并行算法[J]. 计算机应用研究, 2013, 30(3): 814-816. Li Zhengfu, Wang Xicheng, Guo Quan. Parallel algorithm for grid generation of receptor score under CUDA[J]. Computer Application Research, 2013, 30(3): 814-816. DOI:10.3969/j.issn.1001-3695.2013.03.044 |
| [14] |
Vakser I A. Protein-protein docking:From interaction to interactome[J]. Biophysical Journal, 2014, 107(8): 1785-1793. DOI:10.1016/j.bpj.2014.08.033 |
| [15] |
Xu Y, Shen J, Luo X, et al. How does huperzine a enter and leave the binding gorge of acetylcholinesterase? Steered molecular dynamics simulations[J]. Journal of the American Chemical Society, 2003, 125(37): 11340-11349. DOI:10.1021/ja029775t |
| [16] |
Lensink M F, Wodak S J. Docking, scoring, and affinity prediction in CAPRI[J]. Proteins:Structure, Function, and Bioinformatics, 2013, 81(12): 2082-2095. DOI:10.1002/prot.v81.12 |
| [17] |
Janin J. Protein-protein docking tested in blind predictions:The CAPRI experiment[J]. Molecular Biosystems, 2010, 6(12): 2351-2362. DOI:10.1039/c005060c |
| [18] |
Ma X H, Li C H, Shen L Z, et al. Biologically enhanced sampling geometric docking and backbone flexibility treatment with multiconformational superposition[J]. Proteins-Structure Function and Bioinformatics, 2005, 60(2): 319-323. DOI:10.1002/prot.20577 |
| [19] |
Lam P Y, Jadhav P K, Eyermann C J, et al. Rational design of potent, bioavailable, nonpeptide cyclic ureas as HIV protease inhibitors[J]. Science, 1994, 263(5145): 380-384. DOI:10.1126/science.8278812 |
| [20] |
Tao P, Lai L. Protein ligand docking based on empirical method for binding affinity estimation[J]. Journal of Computer-Aided Molecular Design, 2001, 15(5): 429-446. DOI:10.1023/A:1011188704521 |
| [21] |
Mohan V, Gibbs A C, Cummings M D, et al. Docking:Successes and challenges[J]. Current Pharmaceutical Design, 2005, 11(3): 323-333. DOI:10.2174/1381612053382106 |
 2018, Vol. 33
2018, Vol. 33


