2. 皖南医学院健康大数据挖掘与应用研究中心, 芜湖, 241002;
3. 安徽师范大学计算机与信息学院, 芜湖, 241002;
4. 合肥工业大学计算机与信息学院, 合肥, 230009
2. Research Center of Health Big Data Mining and Applications, Wannan Medical College, Wuhu, 241002, China;
3. School of Computer and Information, Anhui Normal University, Wuhu, 241002, China;
4. School of Computer and Information, Hefei University of Technology, Hefei, 230009, China
肿瘤是目前威胁人类生命的主要疾病之一。从分子生物学的角度来看,肿瘤是由于某些染色体上DNA损伤而引起细胞内基因表达异常,导致细胞生长失控、畸形分化和异常增生的一类复杂基因疾病。肿瘤基因表达谱是指利用基因芯片(即DNA芯片)测定基因在肿瘤组织或正常组织等不同样本中表达水平。研究表明,基因表达谱中与肿瘤疾病密切相关的关键基因(又称为特征基因或信息基因)数量非常少。研究基因表达谱、选取信息基因是从信息学角度出发寻找肿瘤分型与分类的基因标记物以及药物治疗潜在靶点的重要手段,同时也是有效构建肿瘤分类模型的关键[1-4]。
随着DNA微阵列技术的快速发展,人们获得大量的基因表达谱,从而为研究肿瘤的发病机制和临床诊断提供了重要依据。然而,基因表达谱存在高维小样本、噪声大且冗余基因多等显著特点,这给基于基因表达谱的肿瘤分类问题研究带来巨大挑战。Guyon等[5]指出:通过DNA微阵列技术获取的基因表达谱中含有大量与特定疾病冗余或不相关的基因。冗余或无关基因的存在,将导致建立肿瘤分类模型费时费力,不可避免陷入过度拟合现象和维数灾难问题[3]。因此,信息基因的选取问题是基于基因表达谱的肿瘤分类的研究核心和极具挑战性的内容,也是生物医学信息学的研究热点之一[1-10]。
从信息学角度考虑,每个基因代表样本的一个特征,如何衡量样本中每个特征所包含的分类信息,准确评估每个特征对样本分类贡献度的大小,这是有效选取信息基因的关键[9]。通常,一个特征集中包含4种特征:无关特征、冗余特征、弱关联非冗余特征和强关联特征[11]。而最佳特征子集仅仅包含两种特征,即弱关联非冗余特征和强关联特征。信息基因选择(即特征选择)是指从基因表达谱的所有基因中选取一个最佳基因子集,即通过剔除无关基因和冗余基因,选择与分类目标存在高度相关性的信息基因子集,从而建立更精确、更易理解的分类模型。通常,特征选择方法可分为3大类[3-8]:过滤法、封装法和嵌入法,其中过滤方法简单、速度快且与分类器无关,并在高维小样本数据上得到更为广泛的应用。过滤方法以特征相关性测度为基础评价特征或特征集的相关性[12]。特征相关性测度可分为特征-类别相关性(又称C-相关性)测度和特征-特征相关性(又称F-相关性)测度[11]。其中,C-相关性测度通过特征对样本类别的区分能力来评价特征的重要性,如T-检验[13]、F-检验[14]、Fisher判别[15]、信息增益[4]、受试者工作特征(Receiver operating characteristic, RoC)曲线[16]和信噪比(Signal noise ratio,SNR)[5]等;F-相关性测度通常是基于信息论或特征自身的统计特性来评价两个特征的相关程度,如互信息[15]、皮尔森相关系数(Pearson correlation coefficient,PCC)[5, 8]和对称不确定性(Symmetric uncertainty,SU)[11, 17, 18]等,并且这些测度也可用于评价特征与类别的C-相关性。
SU是一种利用信息熵描述的非线性相关测度,用于评估两个非线性随机变量之间的相关程度。Yu等[11]提出一种基于特征相关性的快速过滤(Fast correlation-based filter,FCBF)特征选择算法。FCBF算法是根据SU所定义的C-相关和F-相关指标来剔除不相关和冗余特征。该算法首先采用SU评估每个特征的C-相关性并选择前Top K个相关特征,然后根据利用SU定义的近似Markov blanket剔除其中的冗余特征。SU相关性测度的特点为假设所考察特征与其他特征相互独立,SU值只能反映单个特征与类别或两个特征之间的相关性,忽略其他特征对它们的影响。因此,利用SU评估可以从成千上万个基因中选出较少基因作为侯选基因子集,以大幅降低信息基因的搜索空间,但是却不能完全剔除基因集中冗余基因[9]。
粗糙集理论自Pawlak教授于1982年提出以来,得到了广泛的研究和发展[19-22]。然而,Pawlak粗糙集定义在等价关系基础上,只适合处理离散型数据。邻域粗糙集(Neighborhood rough set,NRS)[23]是对Pawlak粗糙集的改进,可以直接处理连续型数据,避免离散化所带来的信息损失,可以有效地剔除特征集中无关和冗余特征,使得所选取的特征子集能够最大限度地保持原始特征集的分类能力。近年来,邻域粗糙集在生物医学信息学领域受到越来越多的关注和研究,并在肿瘤信息基因选择方面已经取得一些研究成果[4, 24]。为了获得基因数量尽可能少而分类能力尽可能强的一组信息基因,本文针对肿瘤基因表达谱自身的特点,提出一种基于SU和NRS的信息基因选择方法SUNRS。
1 对称不确定性和邻域粗糙集相关知识 1.1 对称不确定性SU是一种基于信息熵定义的非线性相关信息度量[11],可用来揭示两个非线性随机变量之间的相关程度。随机变量X的信息熵H(X)定义为
| $ H\left( X \right) = -\sum\limits_i {P({x_i})} {\rm{lo}}{{\rm{g}}_2}P({x_i}) $ | (1) |
式中:P(xi)表示变量X=xi的概率。
在观测到随机变量Y后,随机变量X的信息熵,即条件熵H(X|Y)定义为
| $ H\left( {X|Y} \right) = -\sum\limits_i {P({y_j})} \sum\limits_i P ({x_i}|{y_j}){\rm{lo}}{{\rm{g}}_2}P({x_i}|{y_j}) $ | (2) |
式中:P(yj)表示随机变量Y=yj的概率;P(xi|yj)表示在随机变量Y=yj条件下随机变量X=xi的概率,称为后验概率。
在观测到随机变量Y后,随机变量X的信息熵减少的信息量,即信息增益IG(X|Y)定义为
| $ IG\left( {X|Y} \right) = H\left( X \right) - H\left( {X|Y} \right) $ | (3) |
由式(3)可知,如果变量X和Y不相关,则信息增益IG(X|Y)=0,否则IG(X|Y)>0,IG(X|Y)越大,变量X和Y相关性越强;如果IG(X|Y)>IG(Z|Y),则变量Y和X之间相关性大于变量Y和Z之间相关性。因此,可以用IG(X|Y)来定量评价两个变量之间的相关性。但是,IG(X|Y)结果受到变量单位和变量值的影响,因此需要进一步同质化[11]。
对称不确定性SU(X, Y)是一种规范化的信息增益,SU(X, Y)定义为
| $ {\rm{SU}}\left( {X, Y} \right) = 2\left[{\frac{{IG\left( {X|Y} \right)}}{{H\left( X \right) + H\left( Y \right)}}} \right] $ | (4) |
由式(4)可知,对称不确定性SU(X, Y)满足:0≤SU(X, Y)≤1,当SU(X, Y)=0时,表示两个随机变量X和Y是相互独立的;当SU(X, Y)=1时,表示两个随机变量X和Y是完全相关的。利用SU作为相关性度量,特征选择过程可以通过考虑C-相关(特征与类别的相互关系)和F-相关(特征之间的相互关系)来完成。
文献[11]提出一种利用SU指标剔除不相关和冗余特征的FCBF算法。该算法的基本思想是根据SU所定义的C-相关和F-相关,从原始特征集中剔除C-相关值小于给定阈值的特征,然后再对剩余的特征进行冗余分析。也就是说,如果特征与类别之间的C-相关性较低,则该特征将作为不相关特征消除;如果两个特征之间的F-相关性较强,并且超过它们与类别之间的C-相关性,则认为这两个特征相互冗余,将其中与类别相关性较差的特征作为冗余特征消除。
1.2 邻域粗糙集为了解决Pawlak粗糙集不能直接处理连续型数据的问题,胡清华等[23]在Pawlak粗糙集理论和邻域关系的基础上,提出了邻域粗糙集模型,该模型可以直接处理连续型数据,避免离散化所带来的信息损失。给定分类学习任务〈U, C∪D〉,U={x1, x2, …, xn}是所有对象构成的样本集,C={a1, a2, …, am}是描述样本特征的条件属性集,D={d1, d2, …, dp}是描述样本类别的决策属性集。下面给出邻域粗糙集模型的相关概念和性质。
给定实数空间上的非空有限集合U={x1, x2, …, xn},δ≥0,则对于任意样本xi∈U,B⊆C,xi在属性空间B上的δ邻域δB(xi)定义为
| $ {\delta _B}({x_i}) = \{ {x_j}|{x_j} \in U, {\Delta _B}({x_i}, {x_j}) \le \delta \} $ | (5) |
式中:ΔB(xi, xj)是U上的距离函数,满足ΔB(xi, xj)≥0。
在实际应用中,常见的距离度量是欧氏距离计算函数
| $ {\Delta _B}({x_i},{x_j}) = {\left( {\sum\limits_{k = 1}^n {{{\left( {f\left( {{x_i},{a_k}} \right) - f\left( {{x_j},{a_k}} \right)} \right)}^2}} } \right)^{1/2}} $ | (6) |
式中ak∈B,f(x, ak)为样本x在属性ak上的取值。
论域中所有样本的δ邻域形成了论域的粒化,邻域粒子簇导出的邻域关系构成了论域空间中的邻域决策系统。给定分类学习任务〈U, C∪D〉,设N是由C产生的一簇邻域关系,称〈U, C∪D, N 〉为邻域决策系统[4, 23]。
给定邻域决策系统〈U, C∪D, N 〉,设决策属性集D将论域U划分为n个等价类:U1, U2, …, Un,NB为条件属性子集B⊆C生成U上的邻域关系,则D关于B的δ-邻域下近似、δ-邻域上近似和δ-邻域边界分别定义为
| $ \underline {{N_B}} D = \{ \underline {{N_B}} {U_1}, \underline {{N_B}} {U_2}, \ldots, \underline {{N_B}} {U_n}\} $ | (7) |
| $ \overline {{N_B}} D = \{ \overline {{N_B}} {U_1}, \overline {{N_B}} {U_2}, \ldots, \overline {{N_B}} {U_n}\} $ | (8) |
| $ BN\left( D \right) = \overline {{N_B}} D - \underline {{N_B}} D $ | (9) |
式中:
邻域粗糙集中δ-邻域下近似、δ-邻域上近似和δ-邻域边界的大小不仅与分类问题的样本特征空间有关,而且与分析的信息粒度(即邻域δ取值)有关。邻域δ取值的大小反映了在不同粗细粒度下区分对象,决定了分类边界区训练样本数,因此邻域δ是影响邻域粗糙集模型性能的关键因素。通常,邻域δ取值与研究对象有关,可通过实验进行观察得到。
给定邻域决策系统〈U, C∪D, N 〉,决策属性集D对条件属性子集B⊆C的依赖度定义为
| $ {\gamma _B}\left( D \right) = \frac{{\left| {{\rm{PO}}{{\rm{S}}_B}\left( D \right)} \right|}}{{\left| U \right|}} = \frac{{\left| {\underline {{N_B}} D} \right|}}{{\left| U \right|}} $ | (10) |
给定邻域决策系统〈U, C∪D, N 〉,若B⊆C满足:(1)γB(D)=γC(D);(2)对于任意a∈B存在γB-a(D) < γB(D),则称B是C的一个相对约简。
给定邻域决策系统〈U, C∪D, N 〉,B⊆C,a∈C-B,则条件属性a关于条件属性子集B的重要度定义为
| $ {\rm{SIG}}\left( {a, D, B} \right) = {\gamma _{B \cup a}}\left( D \right){\gamma _B}\left( D \right) $ | (11) |
基于基因表达谱的肿瘤自动检测与分类的一个关键目标就是利用尽可能少的信息基因以获取尽可能高的肿瘤分类精度[24-28]。事实上,仅利用一种信息基因选择方法很难获取满足这一目标的信息基因子集。通常,信息基因子集选取可分为两个阶段[2]:首先利用过滤方法从高维基因数据中获取一定数目的基因作为侯选基因子集,从而大幅缩小信息基因的搜索空间;然后再进一步利用Wrapper方法优选出满足目标的信息基因子集。通过基因排序法选取侯选基因子集是比较常用的一种方法,即依据某种特征相关性测度对所有基因按其C-相关度由高到低进行排序,最后选择Top K个基因作为侯选基因(K通常取50~200)[2]。
通常基因排序法获取的侯选基因是强关联基因,但极有可能选取与之强关联的其他基因作为信息基因,从而产生一些冗余基因。过多的冗余基因容易导致基因子集规模较大而分类精度下降等问题。为了有效消除冗余基因,一些研究者首先使用基因排序方法获取候选基因子集,然后结合Wrapper方法消除冗余,在一定程度上解决了冗余基因带来的缺陷[2, 8, 24]。但是,如果直接采用某种机器学习算法来评估候选基因子集,致使基因选择与学习模型之间相互依赖,容易导致模型过拟合、泛化性能差以及计算开销高等一系列问题[2, 8]。因此,设计鲁棒高效的信息基因选择方法已成为基于基因表达谱的肿瘤自动检测与分类领域中的研究重点。
针对肿瘤基因表达谱的信息基因选择,本文提出基于SU和NRS的信息基因(即特征)选择方法SUNRS,能够有效过滤无关基因并剔消除冗余基因。图 1给出信息基因选择方法SUNRS的系统框架。SUNRS信息基因选择方法分为两层:第1层采用FCBF算法,利用SU指标评估来剔除不相关和冗余基因,得到侯选信息基因集;第2层利用邻域粗糙集模型对侯选信息基因子集进行基因约简,进一步消除冗余基因,获取较优的目标信息基因集。
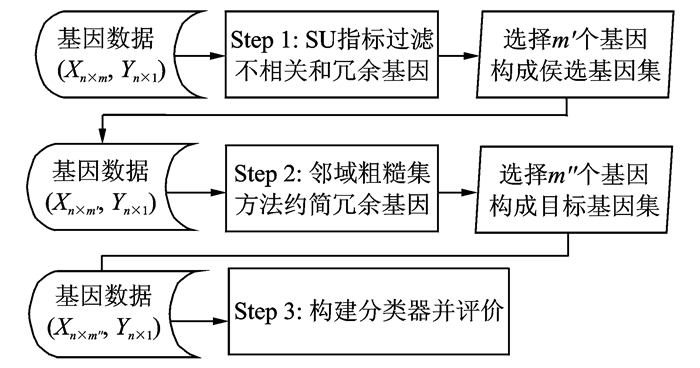
|
图 1 信息基因选择SUNRS方法 Fig. 1 SUNRS for informative gene selection |
假设基因表达谱数据集(X, Y)包含n个样本,m个基因,SUNRS基因选择方法的具体步骤如下:
Step 1:利用SU指标评估信息基因,过滤不相关和冗余基因,从m个基因中获取m′个基因,构成侯选信息基因集;
Step 2:利用邻域粗糙集对m′个信息基因进行基因约简,剔除冗余基因,得到m″个信息基因,构成较优的目标信息基因集;
Step 3:根据目标信息基因集,构建分类模型并评价。
基于对称不确定性和邻域粗糙集的信息基因选择算法SUNRS描述如下。
输入:基因表达谱样本集U={x1, x2, …, xm },样本基因集G={f1, f2, …, fn},样本类别D={Class},C-相关性阈值β,基因邻域参数δ及重要度下限参数λ。
输出:约简后的目标基因集Gred
(1) Glist=∅;//初始化侯选基因集
(2) For i=1 to n do
(3) 计算基因fi与类别Class的C-相关SU(fi, c);
(4) 如果SU(fi, c)>β,则Glist=Glist∪{fi};
(5) End
(6) 按照SU(fi, c)值对Glist中基因进行降序排序;
(7) For i=1 to |Glist| do
(8) 从Glist中取出第i个基因fi;
(9) For j=i+1 to |Glist| do
(10) 从Glist中取出第j个基因fj;
(11) 如果SU(fi, fj)>SU(fj, c),则Glist=Glist-{fi};
(12) End
(13) End
(14) Gred=∅;//初始化目标基因集
(15) For i=1 to |Glist-Gred| do
(16) 计算γGred∪{fi}(D)=|POSGred∪{fi}(D)|/|U|;
(17) 计算SIG(fi, D, Gred)=γGred∪{fi}(D)-γGred(D);
(18) End
(19)选择fk,满足:SIG(fk, D, Gred)=
(20)若SIG(fk, D, Gred)≤λ,则Gred=Gred∪{fk},U=U-POSGred∪{fk}(D),返回(15);否则,输出较优的目标基因集Gred,结束。
在SUNRS算法中,步骤(2~6)根据SU计算每个基因的C-相关值并根据阈值β选择相关特征,然后按C-相关值大小排序,获得初步侯选基因集Glist;步骤(7)~(13)根据以SU定义的近似Markov blanket剔除Glist中的冗余基因,得到侯选基因集Glist,其中根据SU定义的近似Markov blanket:基因fi是基因fj的近似Markov blanket,则SU(fi, c)≥SU(fj, c)且SU(fi, fj)>SU(fj, c);步骤(14~20)根据邻域粗糙集的属性约简方法,剔除Glist中的噪声冗余基因,得到较优的目标基因集Gred。
SU指标假设所考察基因与其他基因相互独立,SU值只能反映单个基因与类别或两个基因之间的相关性,忽略其他基因对它们的影响。因此,利用FCBF算法不能完全消除侯选基因集中冗余基因。SUNRS方法利用邻域粗糙集约简算法进一步剔除侯选基因集上冗余基因,可获得基因数目更少的目标信息基因集,同时能够最大限度地保持与原侯选基因集具有相同的分类信息。
3 实验结果与分析 3.1 实验数据和实验环境为了验证本文所提信息基因选择算法SUNRS的有效性,在5个公开基因表达谱,即结肠癌(Colon)、前列腺癌(Prostate)、淋巴癌(Lymphoma)、白血病(Leukemia)和肺癌(Lung)上进行系列仿真实验。上述基因表达谱可从http://datam.i2r.a-star.edu.sg/datasets/krbd/免费下载,其详细描述如表 1所示。
| 表 1 基因表达谱实验数据集描述 Tab. 1 Description for experimental datasets of gene expression profile |
本文实验中所采用的PC机配置为Intel奔腾双核处理器G645,主频2.90 GHz,内存2 GB和Windows XP操作系统。所有实验均在Weka 3.7.13+Matlab 2012a中实现和完成,利用Weka软件工具在各个基因选择方法选取的目标基因集上构建4种分类模型:即决策树C4.5、随机森林、支持向量机(Support vector machine, SVM)和K-近邻(K-nearest neighbor, KNN),并且各个分类模型的泛化性能均采用留一交叉验证(Leave-one-out cross validation, LOOCV)方法进行评价[3]。
数据预处理是信息基因选择的首要步骤。为了消除不同量纲对实验结果的影响,实验过程中,对基因表达谱进行标准化预处理(均值为0,方差为1),并对各个分类模型的主要参数进行设置,其中决策树C4.5算法中修剪置信因子设为0.25,随机森林中生成树个数(numTrees)设为10,SVM中核函数设为多项式核函数,KNN中邻居数目K设为10[3]。
3.2 实验结果分析本文实验步骤可分为两步:(1)采用FCBF算法,按照对称不确定性SU评估,剔除无关及冗余基因,获取候选信息基因集;(2)在信息基因的候选集基础上,采用邻域粗糙集属性约简方法[4]进一步消除冗余基因,以获得较优的目标信息基因集。
首先,对以上Colon,Prostate,Lymphoma,Leukemia和Lung等5个基因表达谱样本集通过对称不确定性SU定义的C-相关剔除无关基因和F-相关去除冗余基因,最终获得候选的信息基因集数目分别为14,77,73,51和128。然后,利用邻域粗糙集的属性约简方法进一步剔除候选信息基因集中的冗余基因。实验过程中,对基因邻域参数δ及重要度下限参数λ的设置进行对比和优化[4]。经过一系列实验对比,本文实验中Colon,Prostate,Lymphoma,Leukemia和Lung等5个基因表达谱的基因邻域参数δ取值分别为0.26,0.75,1.1,0.9和1.1;参数λ取值越小越好,本文实验中基因重要度下限参数λ取值均为0.000 1。
为了验证本文算法SUNRS选择的目标信息基因集在分类性能上优于其他方法,实验中采用决策树C4.5算法、随机森林Random forest、支持向量机SVM以及K-最近邻KNN等4种分类器来评估几个基因选择算法所选择基因的分类性能(本文选取4种分类器的最高分类精度作为最终的分类性能),并利用LOOCV来评估分类器的泛化性能。
表 2显示在每个原始基因表达谱样本集上和不同信息基因选择算法新获得的每个基因表达谱样本集上的实验结果。在表 2中,ODP[3]表示为原始数据处理(Original data processing,ODP)方法,即在原始基因表达谱样本集上分类建模;SNR表示为只采用信噪比指标(Signal noise ratio,SNR)基因排序法[5],选择Top 100个基因;SNR+Lasso表示为先用SNR方法选择Top 100个基因,再进一步采用Lasso方法[24]剔除冗余基因;SNR+ILasso表示为先用SNR方法选择Top 100个基因,再进一步采用迭代Lasso(Iterative Lasso,ILasso)方法剔除冗余基因[3];FCBF表示为只采用对称不确定性SU指标剔除不相关和冗余基因的FCBF方法[11];SUNRS表示为本文算法即采用基于对称不确定性SU与邻域粗糙集的信息基因选择方法。此外,为便于观察和分析实验结果,表 2中的粗体值表示:在不同基因选择方法选取的目标基因集中,该方法选取的目标基因集最优,即其最优分类性能最高或基因数目最少。
| 表 2 实验对比不同基因选择算法在5个基因表达谱上的最优分类性能(%)和基因数目 Tab. 2 Experimental comparison of gene number and optimal classification performance (%) with different gene selection algorithms on different gene expression profiles |
以下从分类精度和信息基因数量两个方面进行分析。由表 2可知,采用ODP方法直接在原始基因集上分类建模,可以获得较高的分类精度,但是信息基因数量规模过于庞大;SNR方法[5]获取Top 100个基因,分类性能相对较好,但仍存在一些冗余基因;SNR+Lasso方法[3]可有效地消除无关基因,但极有可能将相关性较强且互为冗余的基因误认为是信息基因,从而导致选取的信息基因数仍然过多且分类精度一般;SNR+ILasso方法[3]可获取较少的信息基因集,同时分类性能相对较好;FCBF方法[11]利用SU所定义的F-相关和C-相关指标删除大量无关、冗余基因,获取较少的信息基因集和较高的分类性能,但选取的基因集中仍包含一定数量的冗余基因,分类性能有待提高;SUNRS方法采用SU所定义的F-相关和C-相关指标删除大量无关、冗余基因,然后利用邻域粗糙集再一步约简冗余基因,获得的目标信息基因集不仅基因数目最少,而且具有更好的分类泛化性能。
综合实验结果分析可知,本文所提的SUNRS方法能够选择数量最少的信息基因,并且在分类性能上均不低于其他5种信息基因选择方法,进一步验证了SUNRS方法能够剔除无关基因和冗余基因,选取信息含量较高的强关联基因和弱关联非冗余基因,从而避免基因表达谱具有高维小样本等特点而产生的过度拟合现象以及维数灾难问题[2, 3, 8, 9],提高了模型的分类精度和泛化能力。
4 结束语随着DNA微阵列技术的发展,采用基因表达谱对肿瘤样本进行检测与分类已经成为生物医学信息学的一个重要研究领域[27, 28]。但是,由于目前的基因表达谱具有高维小样本、高噪声和高冗余等特点,促使肿瘤分类检测问题成为生物医学信息学领域研究的一个挑战性工作。针对肿瘤基因表达谱,如何选择数目尽可能少且分类能力尽可能强的信息基因是提高肿瘤分类性能的关键任务一个。本文以肿瘤基因表达谱为研究对象,提出了一种新颖的肿瘤分类信息基因选择SUNRS方法,即基于SU和NRS的信息基因选择方法。实验结果表明,本文提出的信息基因选择方法SUNRS能够选取基因数量少且分类能力较强的目标信息基因集,解决了具有高维小样本特点且普遍存在大量冗余、噪声基因的肿瘤基因表达谱分类问题,进一步提高了肿瘤分类模型的分类准确度和泛化能力。另外,在实际应用中SUNRS方法还存在若干问题有待解决,如FCBF算法中C-特征相关性阈值β、邻域粗糙集的邻域参数δ及重要度下限参数λ在信息基因选择过程中自动寻优确定等问题,都有待进一步研究。
| [1] |
Mohamad M S, Omatu S, Deris S, et al. A modified binary particle swarm optimization for selecting the small subset of informative genes from gene expression data[J]. IEEE Transactions on Information Technology in Biomedicine, 2011, 15(6): 813-822. DOI:10.1109/TITB.2011.2167756 |
| [2] |
王树林, 王戟, 陈火旺, 等. 肿瘤信息基因启发式宽度优先搜索算法研究[J]. 计算机学报, 2008, 31(4): 636-649. Wang Shulin, Wang Ji, Chen Huowang, et al. Heuristic breadth first search algorithm for informative gene selection based on gene expression profiles[J]. Journal of Computers, 2008, 31(4): 636-649. |
| [3] |
张靖, 胡学钢, 李培培, 等. 基于迭代Lasso的肿瘤分类信息基因选择方法研究[J]. 模式识别与人工智能, 2014, 27(1): 49-58. Zhang Jing, Hu Xuegang, Li Peipei, et al. Informative gene selection for tumor classification based on iterative Lasso[J]. Pattern Recognition and Artificial Intelligence, 2014, 27(1): 49-58. |
| [4] |
徐久成, 李涛, 孙林, 等. 基于信噪比与邻域粗糙集的特征基因选择方法[J]. 数据采集与处理, 2015, 30(5): 973-981. Xu Jiucheng, Li Tao, Sun Lin, et al. Feature gene selection based on SNR and neighborhood rough set[J]. Journal of Data Acquisition and Processing, 2015, 30(5): 973-981. |
| [5] |
Golub T R, Slonim D K, Tamayo P, et al. Molecular classification of cancer:Class discovery and class prediction by gene expression monitoring[J]. Science, 1999, 286(10): 531-537. |
| [6] |
Kar S, Sharma K D, Maitra M. Gene selection from microarray gene expression data for classification of cancer subgroups employing PSO and adaptive K-nearest neighborhood technique[J]. Expert Systems with Applications, 2015, 42(1): 612-627. DOI:10.1016/j.eswa.2014.08.014 |
| [7] |
Chen K H, Wang K J, Tsai M L, et al. Gene selection for cancer identification:A decision tree model empowered by particle swarm optimization algorithm[J]. BMC Bioinformatics, 2014, 15(1): 49. DOI:10.1186/1471-2105-15-49 |
| [8] |
谢娟英, 高红超. 基于统计相关性与K-means的区分基因子集选择算法[J]. 软件学报, 2014, 25(9): 2050-2075. Xie Juanying, Gao Hongchao. Statistical correlation and K-means based distinguishable gene subset selection algorithms[J]. Journal of Software, 2014, 25(9): 2050-2075. |
| [9] |
叶明全, 高凌云, 伍长荣, 等. 基于对称不确定性和SVM递归特征消除的信息基因选择方法[J]. 模式识别与人工智能, 2017, 30(5): 429-438. Ye Mingquan, Gao Lingyun, Wu Changrong, et al. Informative gene selection method based on symmetric uncertainty and SVM recursive feature elimination[J]. Pattern Recognition and Artificial Intelligence, 2017, 30(5): 429-438. |
| [10] |
刘金勇, 郑恩辉, 陆慧娟. 基于聚类和微粒群优化的基因选择方法[J]. 数据采集与处理, 2014, 29(1): 83-89. Liu Jinyong, Zheng Enhui, Lu Huijuan. Gene selection based on clustering method and particle swarm optimization[J]. Journal of Data Acquisition and Processing, 2014, 29(1): 83-89. |
| [11] |
Yu L, Liu H. Efficient feature selection via analysis of relevance and redundancy[J]. Journal of Machine Learning Research, 2004, 5(4): 1205-1224. |
| [12] |
钱宇华, 成红红, 梁新彦, 等. 大数据关联关系度量研究综述[J]. 数据采集与处理, 2015, 30(6): 1147-1159. Qian Yuhua, Chen Honghong, Liang Xinyan, et al. Review for variable association measures in big data[J]. Journal of Data Acquisition and Processing, 2015, 30(6): 1147-1159. |
| [13] |
Jeffery I B, Higgins D G, Culhane A C. Comparison and evaluation of methods for generating differentially expressed gene lists from microarray data[J]. BMC Bioinformatics, 2006, 7(10): 359. |
| [14] |
Ding C, Peng H. Minimum redundancy feature selection from microarray gene expression data[J]. Journal of Bioinformatics and Computational Biology, 2003, 3(2): 185-205. |
| [15] |
Cai Ruichu, Hao Zhifeng, Yang Xiaowei, et al. A new hybrid method for gene selection[J]. Pattern Analysis & Applications, 2011, 14(1): 1-8. |
| [16] |
Mamitsuka H. Selecting features in microarray classification using ROC curves[J]. Pattern Recognition, 2006, 39(12): 2393-2404. DOI:10.1016/j.patcog.2006.07.010 |
| [17] |
Kannan S, Ramaraj N. A novel hybrid feature selection via symmetrical uncertainty ranking based local memetic search algorithm[J]. Knowledge-Based Systems, 2010, 23(6): 580-585. DOI:10.1016/j.knosys.2010.03.016 |
| [18] |
Jiang S Y, Wang L X. Efficient feature selection based on correlation measure between continuous and discrete features[J]. Information Processing Letters, 2016, 116: 203-215. DOI:10.1016/j.ipl.2015.07.005 |
| [19] |
Pradipta Maji, Sushmita Paul. Rough set based maximum relevance maximum significance criterion and gene selection from microarray data[J]. International Journal of Approximate Reasoning, 2011, 52(8): 408-426. |
| [20] |
Ye Mingquan, Wu Xindong, Hu Xuegang, et al. Anonymizing classification data using rough set theory[J]. Knowledge-Based Systems, 2013, 43(1): 82-94. |
| [21] |
Ye Mingquan, Wu Xindong, Hu Xuegang, et al. Multi-level rough set reduction for decision rule mining[J]. Applied Intelligence, 2013, 39(3): 642-658. DOI:10.1007/s10489-013-0439-3 |
| [22] |
叶明全, 胡学钢, 胡东辉, 等. 基于属性值分类的多层次粗糙集模型[J]. 模式识别与人工智能, 2013, 26(5): 481-491. Ye Mingquan, Hu Xuegang, Hu Donghui, et al. A multi-level rough set model based on attribute value taxonomies[J]. Pattern Recognition and Artificial Intelligence, 2013, 26(5): 481-491. |
| [23] |
胡清华, 于达仁, 谢宗霞. 基于邻域粒化和粗糙逼近的数值属性约简[J]. 软件学报, 2008, 19(3): 640-649. Hu Qinghua, Yu Daren, Xie Zongxia. Numerical attribute reduction based on neighborhood granulation and rough approximation[J]. Journal of Software, 2008, 19(3): 640-649. |
| [24] |
Wang Shulin, Li Xueling, Zhang Shanwen, et al. Tumor classification by combining PNN classifier ensemble with neighborhood rough set based gene reduction[J]. Computers in Biology and Medicine, 2010, 40(2): 179-189. DOI:10.1016/j.compbiomed.2009.11.014 |
| [25] |
Wang Yuhang, Makedon FS, Ford JC, et al. HykGene:A hybrid approach for selecting marker genes for phenotype classification using microarray gene expression data[J]. Bioinformatics, 2005, 21(8): 1530-1537. DOI:10.1093/bioinformatics/bti192 |
| [26] |
Zheng Songfeng, Liu Weixiang. An experimental comparison of gene selection by Lasso and dantzig selector for cancer classification[J]. Computers in Biology and Medicine, 2011, 41(11): 1033-1040. DOI:10.1016/j.compbiomed.2011.08.011 |
| [27] |
张丽娟, 李舟军. 微阵列数据癌症分类问题中的基因选择[J]. 计算机研究与发展, 2009, 46(5): 794-802. Zhang Lijuan, Li Zhoujun. Gene selection for cancer classification in microarray data[J]. Journal of Computer Research and Development, 2009, 46(5): 794-802. |
| [28] |
Zou Quan, Zeng Jiancang, Cao Liujuan, et al. A novel features ranking metric with application to scalable visual and bioinformatics data classification[J]. Neurocomputing, 2016, 173(Part 2): 346-354. |
 2018, Vol. 33
2018, Vol. 33

